检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
2022年11月11日,金沙集团wwW3354CC伊成器课题组和合作者联合在Nature Biotechnology杂志上在线发表了题为“Re-engineering the adenine deaminase TadA-8e for efficient and specific CRISPR-based cytosine base editing”的研究成果。该项研究通过在TadA-8e腺嘌呤脱氨酶中引入N46L突变,成功将其转变为专一且高效的胞嘧啶脱氨酶,并以此为基础开发了新型的碱基编辑器Td-CGBE与Td-CBEs。

相比于传统的基于AID/APOBEC家族的CGBE/CBE碱基编辑器,Td-CGBE/CBEs编辑器在编辑活性与之类似的情况下,展现出更窄的编辑窗口和更低的插入、缺失副产物等编辑特性。同时,利用之前开发的全基因组、无偏好性的脱靶检测技术Detect-seq,对Td-CBEs编辑器进行了脱靶评估。评估发现,Td-CBEs在全基因组水平所造成的脱靶位点要远少于同等编辑活性的传统CBEs;虽然Td-CBEs与传统碱基编辑器BE4max基于不同的脱氨酶构建,但Td-CBEs相比于BE4max不会产生新的脱靶位点。值得注意的是,Td-CBEs也并不会产生之前课题组发现的两类新型脱靶编辑(out-of-protospacer edits与target-strand edits)。
以上结果表明,Td-CBEs是一种较传统CBEs更为安全的碱基编辑器,为未来遗传疾病的基因治疗带来了更大的可能。同时,Detect-seq作为一种平台技术,可以为开发更安全、高效的基因编辑工具提供强有力的支持与帮助。
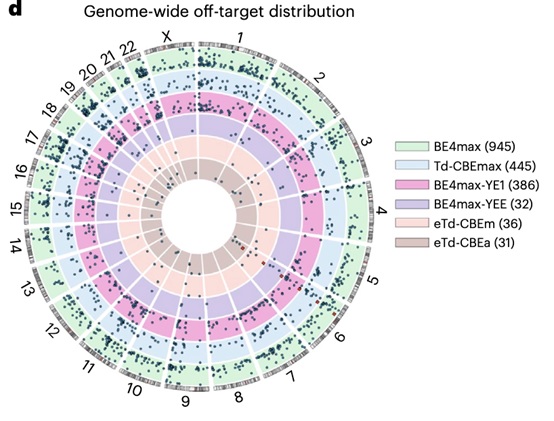
图- 2 利用Detect-seq技术对Td-CBEs进行脱靶检测与评估
北京大学生科院伊成器教授和华东师范大学李大力教授,刘明耀教授为本文通讯作者。华东师范大学金沙集团wwW3354CC研究生陈亮,朱碧云,汝高盟,以及北京大学生科院博士后孟浩巍,博士研究生闫永昌为本论文的共同第一作者。北京大学前沿交叉学院雷芷芯博士,博士研究生乌浩在本研究中做出了重要贡献。该工作得到科技部重点研发计划和国家自然科学基金等项目资助。北京大学高性能计算平台,金沙集团wwW3354CC仪器中心及凤凰工程等多个平台对本项目提供了重要的技术支撑。
原文链接:https://www.nature.com/articles/s41587-022-01532-7