检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
细胞中的生物过程涉及到DNA、RNA、蛋白质等多种不同层次的调控,它们相互影响,共同发挥作用,因此,整合不同组学数据对应的多模态信息是全面表征细胞生理/病理状态的前提与关键。
近年来单细胞多组学技术的发展使得生物学家可以同时在一个细胞中测得不同的模态/组学信息(SHARE-seq, Sci-Car, InCite-seq, 10X multiome),有了对同一个系统的不同模态的认识,可以进一步加深对重要生命过程的理解,比如疾病,胚胎发育。但是这些多组学技术相较于之前的单组学技术,实际应用更困难,花费成本更高,得到的数据质量也更差。因此开发一种计算方法,来利用这些单细胞的多组学数据作为监督信号,整合目前已有的大量高质量的单模态数据将会对这个领域提供巨大的帮助(图1)。
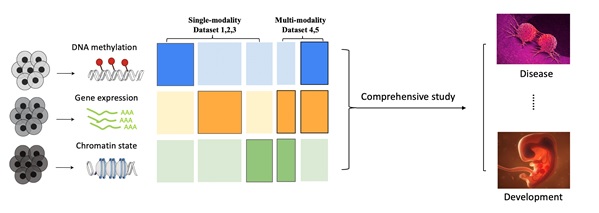
图1:在单细胞组学研究中的跨模态表征学习
针对这一问题,高歌团队提出了跨模态表征学习下的交联表征学习框架(Cross-linked Unified Embedding),相关论文被人工智能领域顶会NeurIPS 2022 接受,并获邀请做专题亮点报告(oral presentation) ,相关论文和代码均已开源。
单细胞多模态数据整合的常见范式是通过每个模态特有的编码器,将来自不同特征空间的数据投影到低维空间中,再通过学习匹配函数 (alignment methods) ,利用来自多组学技术的配对监督信号将模态特异的低维表征整合到一起。但是这些方法都有一个共同的局限,他们没有考虑到不同模态之间的分辨率是不同的,比如免疫细胞在表面蛋白这个模态上拥有更细致的表征,但是在整体基因表达上差异相对较小。因此在整合过程中,低分辨率的基因表达空间会影响高分辨的蛋白空间,从而损失这些模态特异性的信息。换言之,这些不同模态之间会相互掣肘,而不是共同促进。
为了解决这个问题,CLUE 引入模态特异的表征子空间,对每个模态有一个对应的子空间来学习对应模态的信息,从而消除不同模态之间由分辨率不同产生的互相限制。与此同时,CLUE进一步针对不同模态使用自模态编码器(self-encoder),来学习单个模态中的原始信息,利用跨模态编码器(cross-encoder)学习不同模态之间的信息,进而通过多模态之间的映射将这些来自不同模态的表征整合到一起(图 2)。
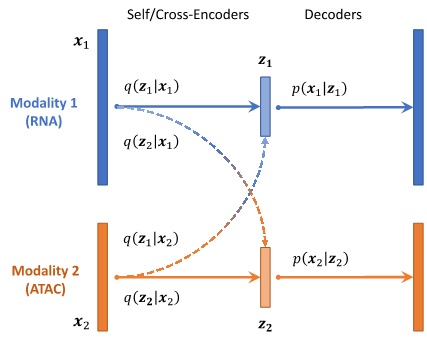
图2:CLUE模型框架示意图
除此之外, CLUE也引入了对抗学习(Adversarial learning)以此消除不同模态之间的表征差异,以及借助多组学的监督信号来优化配对多模态表征的均方误差(mean square error),从而进一步提高整合的准确性。
在首届NeurIPS多模态单细胞数据整合竞赛中,CLUE在包括单细胞染色质开放组/转录组/表面蛋白组在内的所有整合类别跨模态整合中均以压倒性优势取得了冠军(图3)。与此同时,CLUE在于MultiVI, Cobolt, Bridge-integration等尚未参加比赛的整合方法的比较中也取得了最优的表现。
CLUE 在单细胞多组学中的相关模型已经整合入高歌课题组此前开发的基于Python的开源软件包 GLUE (https://github.com/gao-lab/GLUE)中。值得注意的是,CLUE的设计并不局限于单细胞多组学数据,原则上可以被扩展应用至图像/文本/音频等多种模态领域。
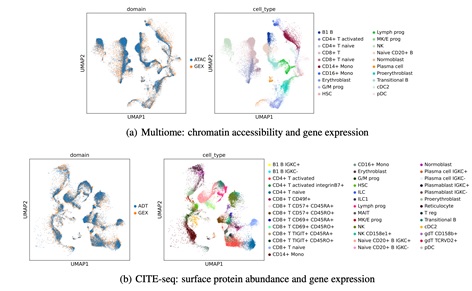
图 3 CLUE 在单细胞染色质开放组,转录组,表面蛋白组上的整合结果
金沙集团wwW3354CC本科生屠鑫明(现华盛顿大学在读博士生),北京大学“博雅”博士后曹智杰博士为该论文共同第一作者,北京大学研究生夏辰睿为共同作者,北京大学/昌平实验室高歌研究员为本文通讯作者,屠鑫明同学现导师华盛顿大学Sara Mostafavi教授为该论文的共同通讯作者。该研究得到了国家重点研发计划、蛋白质与植物基因研究国家重点实验室、北京未来基因诊断高精尖创新中心和昌平实验室的资助。
开源代码: https://github.com/gao-lab/GLUE
论文全文: https://openreview.net/pdf?id=Tfb73TeKnJ-
1. Tu, X*., Zhijie-Cao*, Xia, C., Mostafavi, S. & Gao, G. Cross-Linked Unified Embedding for cross-modality representation learning. in 36th Conference on Neural Information Processing Systems (NeurIPS 2022)
2. Stuart, T., Butler, A., Hoffman, P., Hafemeister, C., Papalexi, E., Mauck, W. M., Hao, Y., Stoeckius, M., Smibert, P. & Satija, R. Comprehensive Integration of Single-Cell Data. Cell 177, (2019).
3. Ma, S., Zhang, B., LaFave, L. M., Earl, A. S., Chiang, Z., Hu, Y., Ding, J., Brack, A., Kartha, V. K., Tay, T., Law, T., Lareau, C., Hsu, Y.-C., Regev, A. & Buenrostro, J. D. Chromatin Potential Identified by Shared Single-Cell Profiling of RNA and Chromatin. Cell 183, 1103-1116.e20 (2020).
4. Cao, J., Cusanovich, D. A., Ramani, V., Aghamirzaie, D., Pliner, H. A., Hill, A. J., Daza, R. M., McFaline-Figueroa, J. L., Packer, J. S., Christiansen, L., Steemers, F. J., Adey, A. C., Trapnell, C. & Shendure, J. Joint profiling of chromatin accessibility and gene expression in thousands of single cells. Science 361, 1380 1385 (2018).
5. Chung, H., Parkhurst, C. N., Magee, E. M., Phillips, D., Habibi, E., Chen, F., Yeung, B. Z., Waldman, J., Artis, D. & Regev, A. Joint single-cell measurements of nuclear proteins and RNA in vivo. Nat Methods 18, 1204–1212 (2021).
6. Janssens, J., Aibar, S., Taskiran, I. I., Ismail, J. N., Gomez, A. E., Aughey, G., Spanier, K. I., Rop, F. V. D., González-Blas, C. B., Dionne, M., Grimes, K., Quan, X. J., Papasokrati, D., Hulselmans, G., Makhzami, S., Waegeneer, M. D., Christiaens, V., Southall, T. & Aerts, S. Decoding gene regulation in the fly brain. Nature 1–7 (2022). doi:10.1038/s41586-021-04262-z
7. Argelaguet, R., Clark, S. J., Mohammed, H., Stapel, L. C., Krueger, C., Kapourani, C.-A., Imaz-Rosshandler, I., Lohoff, T., Xiang, Y., Hanna, C. W., Smallwood, S., Ibarra-Soria, X., Buettner, F., Sanguinetti, G., Xie, W., Krueger, F., Göttgens, B., Rugg-Gunn, P. J., Kelsey, G., Dean, W., Nichols, J., Stegle, O., Marioni, J. C. & Reik, W. Multi-omics profiling of mouse gastrulation at single-cell resolution. Nature 576, 487–491 (2019).
8. Welch, J. D., Kozareva, V., Ferreira, A., Vanderburg, C., Martin, C. & Macosko, E. Z. Single-Cell Multi-omic Integration Compares and Contrasts Features of Brain Cell Identity. Cell 177, (2019).
9. Argelaguet, R., Cuomo, A. S. E., Stegle, O. & Marioni, J. C. Computational principles and challenges in single-cell data integration. Nat Biotechnol 39, 1202–1215 (2021).
10. Lance, C., Luecken, M. D., Burkhardt, D. B., Cannoodt, R., Rautenstrauch, P., Laddach, A., Ubingazhibov, A., Cao, Z.-J., Deng, K., Khan, S., Liu, Q., Russkikh, N., Ryazantsev, G., Ohler, U., participants, N. 2021 M. data integration competition, Pisco, A. O., Bloom, J., Krishnaswamy, S. & Theis, F. J. Multimodal single cell data integration challenge: results and lessons learned. Biorxiv 2022.04.11.487796 (2022). doi:10.1101/2022.04.11.487796
11. Cao, Z.-J. & Gao, G. Multi-omics single-cell data integration and regulatory inference with graph-linked embedding. Nat Biotechnol 1–9 (2022). doi:10.1038/s41587-022-01284-4