检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
自2022年5月疫情爆发以来,猴痘在世界范围内广泛传播,威胁人类健康。今年6月份以来,我国猴痘病例骤增,国家卫健委已将猴痘纳入乙类传染病管理。引发此次疫情的猴痘病毒(monkeypox virus, MPXV)属于痘病毒科(Poxviridae family)正痘病毒属(Orthopoxvirus genus),是一种大型、有包膜、双链DNA病毒。该家族还有两个熟知的成员,痘苗病毒(vaccinia virus)和天花病毒(variola virus)。DNA复制机器尤其是DNA聚合酶是抗正痘病毒药物的重要靶标,该复合物高分辨率结构的解析能够为抗猴痘药物和疫苗的开发提供关键的结构基础。
大部分DNA病毒在宿主细胞核内进行基因组的复制,而包括猴痘病毒在内的痘病毒的基因组复制在宿主胞质中完成,其DNA复制所需的关键蛋白因子主要由病毒基因组编码,包括DNA聚合酶F8(猴痘病毒和痘苗病毒同源蛋白命名有一定差异,本文出现的蛋白因子均按猴痘病毒系统命名)、DNA解旋酶/引物酶E5、尿嘧啶DNA糖苷酶(uracil-DNA glycosidase,UDG)E4、与E4共同组成聚合酶复制持续性因子(processivity factor)异源二聚体的A22以及多功能磷酸化蛋白H5等。F8是DNA聚合酶复合物的核心催化亚基,但只能合成小于10碱基的小片段产物,A22-E4二聚体与F8形成稳定三元复合物,可以增强聚合酶复制的持续性,因此该三元复合物也被认为是痘病毒的DNA聚合酶全酶。此外,UDG E4可以识别并切除DNA中的尿嘧啶单碱基突变并启动DNA修复途径。UDG作为必需因子整合到聚合酶全酶中是痘病毒聚合酶所特有的,这种DNA合成和尿嘧啶碱基搜索/切除两个过程耦合的机制尚不明确(Moss, 2013)。H5是一种多功能蛋白,也是痘病毒DNA复制的必需因子,但针对H5在DNA复制过程中的分子机制还完全不清楚(Boyle et al., 2015; McCraith et al., 2000)。

2023年12月7日,金沙集团wwW3354CC、PKU-THU生命科学联合中心高宁团队在Molecular Cell杂志最新一期发表题为“Structural insights into the assembly and mechanism of monkeypox virus DNA polymerase complex F8-A22-E4-H5”的研究论文,解析了猴痘病毒DNA聚合酶全酶四元复合物不同功能状态的高分辨率结构,阐释了这些病毒复制因子在DNA复制过程中的分子机制。
围绕DNA聚合酶F8和UDG E4的功能,该工作通过在复合物中加入不同DNA底物的方式,捕捉到F8-A22-E4-H5四元复合物不同的构象状态,包括聚合酶在工作过程中为维持复制保真性需要不断转换的经典的“open”和“closed”两种构象状态,以及UDG E4结合特异性底物(包含dU碱基的DNA)的催化后构象。作者对这些复合物样品分别进行了冷冻电镜结构解析,最高分辨率达到2.7 Å(图1)。F8、A22和E4以1:1:1形式存在,而H5以四聚体形式结合在复合物中。A22含有四个结构域,呈U型结构分布。作为脚手架蛋白,A22同时介导了三个功能元件F8、E4和H5的整合,是聚合酶四元复合物组装的核心元件。
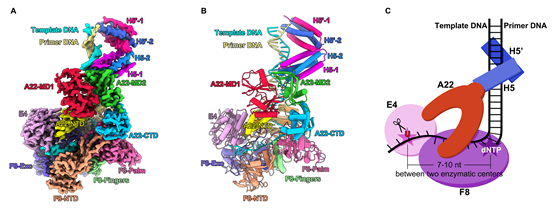
图1,猴痘病毒DNA聚合酶F8-A22-E4-H5四元复合物冷冻电镜结构(A-B)及模式图(C)。
UDG E4结合在单链模板DNA区域,位于聚合酶F8酶活中心上游(图1C)。在添加有E4特异性DNA底物(-7位为尿嘧啶,dU-7)的四元复合物结构中,可以看到U碱基被切除(图2A-B),形成一个无碱基的位点,表明聚合酶中的E4具有UDG酶活性,也再次从结构证明了痘病毒中DNA尿嘧啶单碱基修复和DNA合成过程是耦合在一起的。不同状态的结构分析表明E4和F8的两个活性中心之间具有7-10 nt的间隔(图1C)。同其它UDG类似,以往的E4结构和功能研究主要以dsDNA底物为研究对象。作者首次获得了E4结合有ssDNA的结构,发现E4结合和作用于ssDNA的机制与dsDNA类似(图2B-C)。
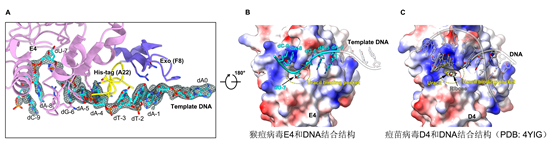
图2,E4和ssDNA(A-B)以及dsDNA(C)底物之间的相互作用比较。
原核生物和真核生物经典的DNA聚合酶中的复制持续性因子PCNA结合在聚合酶活性中心下游的dsDNA区域(图3A),环绕dsDNA防止DNA底物的脱落。在猴痘病毒聚合酶四元复合物结构中,已知的复制持续因子A22-E4结合在活性中心上游的模板ssDNA区域(图1C),在分子机制上与PCNA完全不同。该工作的结构分析认为,A22-E4可能一方面通过E4与ssDNA的结合特性促进聚合酶与DNA底物的结合,另一方面A22、E4以及F8的部分结构域共同组成了一个封闭的环,环绕ssDNA区域防止DNA底物的脱落,从而增加聚合酶的复制可持续性(图1C)。
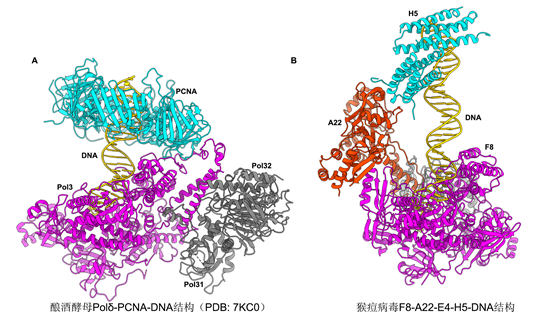
图3,H5四聚体和PCNA在DNA聚合酶复合物中的结合位点比较。
尽管H5是一个必需复制因子,它的分子角色一直不甚清楚。在四元复合物结构中,两个H5二聚体进一步形成四聚体。四聚体通过一个末端与A22互作从而锚定在聚合酶全酶复合物中,同时以半圆形式环绕dsDNA。有趣的是,H5四聚体在痘病毒聚合酶中结合DNA的位置与PCNA结合DNA的位置类似,都位于酶活中心下游的dsDNA区域,表明H5可能也具有促进复制可持续性的功能(图3)。该工作进一步结合生化实验验证了H5在复制过程中的这一功能。
在本论文撰写及同行评议过程中,施一/高福团队、董长江/张郑宇团队、鄢仁鸿团队先后报道了猴痘病毒F8-A22-E4三元复合物的结构(Li et al., 2023; Peng et al., 2023; Xu et al., 2023),展示了三元复合物的亚基组织及其与DNA底物互作的分子细节。这些工作未涉及到此前功能未知的必需复制因子H5,也缺少UDG E4和DNA底物之间互作的结构信息。本项工作通过一系列高分辨率的F8-A22-E4-H5结构和功能实验,揭示了H5四聚体增强聚合酶的持续性的分子功能,证明了E4在聚合酶复合物中具有碱基切除的催化活性。有意思的是,领域的前期工作表明痘病毒聚合酶不具备跨损伤合成活性(translesion synthesis)(Boyle et al., 2011),表明E4在ssDNA模板上切除U碱基后产生的无碱基位点还需要进一步修复,否则将会影响四元复合物中F8所介导的DNA合成,这暗示着病毒完整的复制体还可能包括更多的碱基切除修复通路的因子。总体上,本项工作和其他团队最近发表的相关工作一起为理解痘病毒独特的DNA复制分子机制提供了重要的结构和功能信息。值得一提的是,该论文提供的图片被选为了当期杂志的封面(图4)。

图4,Molecular Cell 当期(Volume 83, Issue 23)的封面图片。
封面图展示了猴痘病毒DNA聚合酶F8在病毒因子A22、E4和H5的帮助下进行DNA复制的分子机制。其中两条螺旋缠绕的树藤分别代表病毒基因组的模板DNA链和新生DNA链;建筑工人代表F8;红色安全带代表A22-E4,安全脚蹬代表H5,强调了A22-E4和H5分别以不同的分子机制增强F8的DNA复制可持续性。
高宁教授和李宁宁副研究员为本文的共同通讯作者,金沙集团wwW3354CC2019级博士研究生王笑涵为本文第一作者,前沿交叉学院2022级博士研究生马靓雯(昌平实验室研究生项目)也参与了这项工作。本研究得到了生命科学联合中心、国家自然科学基金、科技部重点研发计划、昌平实验室的经费支持。这项工作的冷冻电镜样品检查和数据收集主要在北京大学冷冻电镜平台完成,部分数据在水木未来(北京)科技有限公司收集。北京大学高性能计算平台、金沙集团wwW3354CC仪器中心及国家蛋白质基础设施(北大分平台)为本项目提供了重要的技术支撑。
原文链接:https://www.cell.com/molecular-cell/fulltext/S1097-2765(23)00909-7
参考文献:
Boyle, K.A., Greseth, M.D., and Traktman, P. (2015). Genetic Confirmation that the H5 Protein Is Required for Vaccinia Virus DNA Replication. J Virol 89, 6312-6327.
Boyle, K.A., Stanitsa, E.S., Greseth, M.D., Lindgren, J.K., and Traktman, P. (2011). Evaluation of the Role of the Vaccinia Virus Uracil DNA Glycosylase and A20 Proteins as Intrinsic Components of the DNA Polymerase Holoenzyme. J Biol Chem 286, 24702-24713.
Li, Y., Shen, Y., Hu, Z., and Yan, R. (2023). Structural basis for the assembly of the DNA polymerase holoenzyme from a monkeypox virus variant. Sci Adv 9, eadg2331.
McCraith, S., Holtzman, T., Moss, B., and Fields, S. (2000). Genome-wide analysis of vaccinia virus protein-protein interactions. Proc Natl Acad Sci U S A 97, 4879-4884.
Moss, B. (2013). Poxvirus DNA replication. Cold Spring Harb Perspect Biol 5.
Peng, Q., Xie, Y., Kuai, L., Wang, H., Qi, J., Gao, G.F., and Shi, Y. (2023). Structure of monkeypox virus DNA polymerase holoenzyme. Science 379, 100-105.
Xu, Y., Wu, Y., Zhang, Y., Fan, R., Yang, Y., Li, D., Zhu, S., Yang, B., Zhang, Z., and Dong, C. (2023). Cryo-EM structures of human monkeypox viral replication complexes with and without DNA duplex. Cell Res 33, 479-482.